
茶树根系评估新突破:高光谱数据驱动LSTM模型精准预测生物量
应用方向:
在本研究中,高光谱成像技术被应用于茶树扦插苗的生长监测与建模,展现出在农业育种与智能管理中的广阔应用前景。通过对叶片和嫩梢的高光谱反射数据采集,结合深度学习可以在不破坏样本的前提下实现茶苗地上部和根系生物量的快速、精准预测,解决根系难以直接测量的问题,为替代传统耗时且破坏性的称重方法提供了有效途径。因此,高光谱技术不仅能够为茶树优良品种的筛选和扦插苗的生长监测提供可靠手段,还可为苗圃精细化管理和智能化育苗奠定技术基础。
背景:
茶树是全球重要的经济作物,具有庞大的消费市场,但茶苗的繁育与生长受到**气候条件等因素的限制,导致茶树扦插苗生长缓慢、成本高昂,从而制约了优良品种的推广与产业化水平。扦插苗的地上部和根系生物量是衡量其生长发育质量的重要指标,因此,如何快速、准确地监测其生长过程对于提高成活率和苗木管理水平具有重要意义。
传统的人工称重方法在测定茶树扦插苗生物量时具有破坏性且效率低,难以满足高通量、无损检测的需求。近年来,高光谱成像技术因其能够同时获取样品的结构和化学信息,被广泛应用于作物性状监测。已有研究表明,光谱信息与植物的生理、生化特征存在紧密联系,可用于反映植株的养分含量、**水平及生理状态。结合机器学习和深度学习的方法,能够进一步提升高光谱数据的建模精度与稳定性。
因此,该研究提出利用高光谱成像技术结合深度学习方法,对茶树扦插苗的地上部和根系生物量进行快速、无损预测,以解决传统方法耗时、破坏性强的问题,为茶树优良品种的高效筛选和精细化育苗管理提供新的技术途径和数据支撑。
作者信息:丁兆堂;山东省农业科学院
期刊来源:Scientia Horticulturae
研究内容
本研究旨在利用高光谱成像技术结合深度学习模型,实现对茶树扦插苗地上部和根系生物量的快速、无损与精准预测,以解决传统人工称重方法破坏性强、效率低的问题。研究过程中,首先采集不同生长阶段茶树扦插苗叶片与嫩梢的高光谱反射数据,并对光谱数据进行预处理,以去除噪声和散射效应并增强光谱与生理指标的相关性。随后,提取关键光谱特征波段,减少冗余信息并突出与生物量密切相关的变量。在建模环节,利用 Mask R-CNN 网络对扦插苗进行图像与光谱特征融合,构建CNN-GRU生物量预测模型。研究建立了一种高效可靠的检测方法,为茶树优良品种的筛选、扦插苗的生长监测以及智能化育苗管理提供了新的技术途径与数据支撑。
实验设计
茶树扦插苗选用了三个品种:‘玉金香(YJX)’、‘中白1号(ZB)’和‘中茗6号(ZM)’。为了获取不同生长期的茶扦插苗,实验每25天进行一次,共进行了10次。每次试验1个穴盘(含32株扦插苗),共收获扦插苗960株(3个品种× 32株扦插苗× 10个试验)。
为了测定扦插苗的生长情况,对960条扦插苗的枝条和根系进行破坏性分析,并采集高光谱数据,将枝条和根系置于105 ℃烘箱中20 min,然后将烘箱温度调至90 ℃干燥至恒重,*后用电子秤记录重量
本研究采用了高光谱成像技术来监测茶扦插苗从扦插到成长为幼苗过程中生物量的变化(见图1a)。高光谱成像采集系统包括成像光谱相机(Gaia field pro-v10,江苏双利合谱技术有限公司),四个卤素灯、电脑、校正白板、黑色背景。高光谱相机所拍摄图像的光谱范围在可见-近红外波段(391-1010 nm),光谱范围为1101 × 960像素,可测量360个波段的光谱反射率。为了避免光谱相机内部暗电流的影响,提高高光谱图像的信噪比,对获取的原始高光谱图像进行黑白白色校正。利用高光谱相机采集了960株扦插苗的高光谱数据,每幅高光谱图像包含8株扦插苗作为一个模型样本,得到包括120个高光谱图像的总数据集。通过结合成熟叶片和茎叶的光谱信息以及深度学习和机器学习算法,对茎叶和根系的生物量进行了评估(见图1b)。
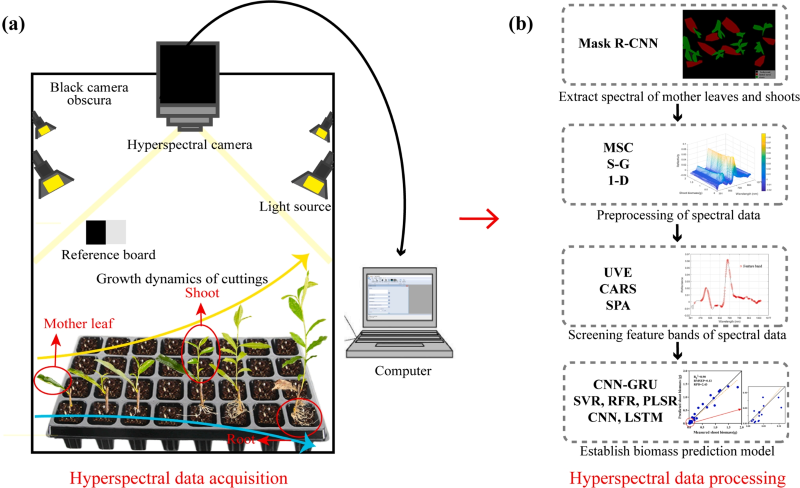
图1. 图像采集与流程图的结合。(a) 图像采集 (b) 数据处理流程图
研究方法
为了增加模型训练的数据量,120张图像通过两种不同的数据增强方法进行处理:旋转(90°、180°和270°)和翻转(水平和垂直),图像数量增加到720张。这些图像通过Labelme软件进行手动标注。首先,对成熟叶片和茎叶区域分别用不同颜色的标签进行标注和分类,其中绿色代表茎叶,红色代表成熟叶片。未标注的区域被视为背景。图2展示了成熟叶片和茎叶的标注图像。随后,将这些标注图像输入到Mask R-CNN中进行训练。

图2. 标注图像 (a) 标注成熟叶片和茎叶的图像;(b) 标注完成的成熟叶片和茎叶图像
Mask R-CNN网络被用于获取茎叶和成熟叶片的面积。图3展示了Mask R-CNN网络的结构图。Mask R-CNN 网络主要分为五个结构:Backbone、区域建议网络(RPN)、感兴趣区域对齐(ROI Align)、边框回归(Box Regression)以及分类与掩膜(Classification and Mask)。通过5折交叉验证将标记的图像划分为训练集和测试集。所采用的学习率为0.001,Epoch为20,Batch大小为1。
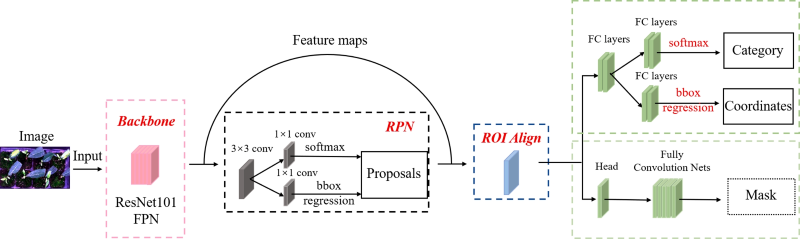
图3. Mask R-CNN的结构图。
由于高光谱采集仪器及环境因素的影响,在成熟叶片和嫩梢的光谱中存在散射效应、随机噪声和系统噪声。因此,本研究对其光谱数据进行了 MSC、S-G 和一阶导数(1-D)预处理。另外为了减少数据计算量并提高后续建模的准确性,本研究采用了连续投影算法(SPA)、竞争自适应重加权采样算法(CARS)和不确定性变量消除算法(UVE)来选择具有代表性的光谱波段作为特征波段。
在模型的建立方面,构建了卷积神经网络与门控循环单元(CNN-GRU)网络模型和传统机器学习模型。CNN-GRU模型网络结构如图4所示。首先,为了更好地提取数据的底层特征,使用CNN来提取数据特征。然后,数据被输入到一个5×5的滤波器中进行卷积。经过4次卷积、平均池化、序列扩展和展平后,数据被输入到GRU网络中。在这里,一维光谱数据与茎叶和根系生物量数据被GRU网络结合,用于回归预测。此外,在CNN网络中,步长为1,填充为“same”(填充值由算法根据卷积核大小内部计算),输入数据通道为1。经过3次门控循环后,预测数据被输入到全连接层,并通过回归器输出。
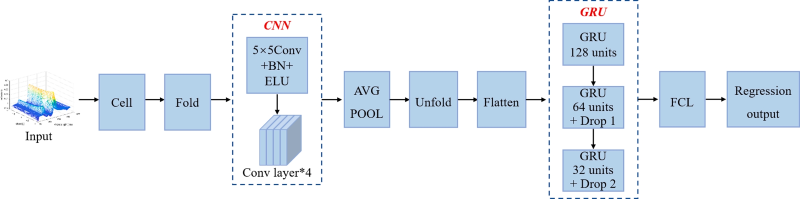
图4. CNN-GRU的结构图。
为了进一步验证CNN-GRU网络的性能,使用了SVR、RFR和PLSR三种机器学习方法和CNN、LSTM两种深度学习方法作为对比模型,CNN和LSTM网络的层数均设置为16层。为了防止模型的过拟合,在训练过程中确定了超参数,支持向量回归机的核函数为多项式核函数,RFR的树的数量为200,PLSR的隐变量为18。在本研究中,用6种方法分别建立了以地上部、成熟叶和地上部、根系生物量为指标的回归模型。
为了进一步保证算法的准确性,本研究采用五重交叉验证,将数据集分成五部分,依次取其中4个作为训练数据,1个作为测试数据,重复五次,然后对结果进行平均,为了更准确地评价回归模型的性能,使用决定系数(R2)、均方根误差(RMSE)、归一化均方根误差(NRMSE)和相对百分比偏差(RPD)来评价模型的性能。为了评估Mask R-CNN模型的光谱信息提取性能,使用精度,召回率和F1得分来评估模型的性能。
结果
通过实验室方法测定了茶扦插苗的茎叶和根系生物量。结果显示,三种品种的扦插苗茎叶萌发时间相同,均在第25天开始萌发。然而,中茗6号(ZM)的生根时间*早,从第125天开始生根;中茗6号的茎叶和根系生长量*大,茎叶生长量约为1.7克,根系生长量约为0.6克。在200–225天期间,茎叶和根系的生长速度*快;中白1号(ZB)的生根时间*晚,从第175天开始生根;玉金香(YJX)的茎叶和根系生长量*小,茎叶生长量约为0.8克,根系生长量约为0.1克。
对成熟叶片和茎叶的分割结果进行了比较和分析,结果显示,Mask R-CNN能够以高精度分割成熟叶片和茎叶。其中,成熟叶片光谱的提取效果优于茎叶光谱,提取精度达到97.8%。茎叶光谱的提取精度为91.5%。成熟叶片和茎叶光谱的提取精度均超过90%。因此,Mask R-CNN模型能够准确且高效地从茶扦插苗图像中提取成熟叶片和茎叶的光谱信息。
对Mask R-CNN模型提取的成熟叶片和枝条原始光谱分别采用MSC、1-D和S-G算法进行预处理,如图5所示,与原始光谱相比,MSC、1-D和S-G联合预处理后光谱曲线的波峰和波谷更加突出,提高了光谱的分辨率和灵敏度,有利于提高后期建立回归模型的准确性。
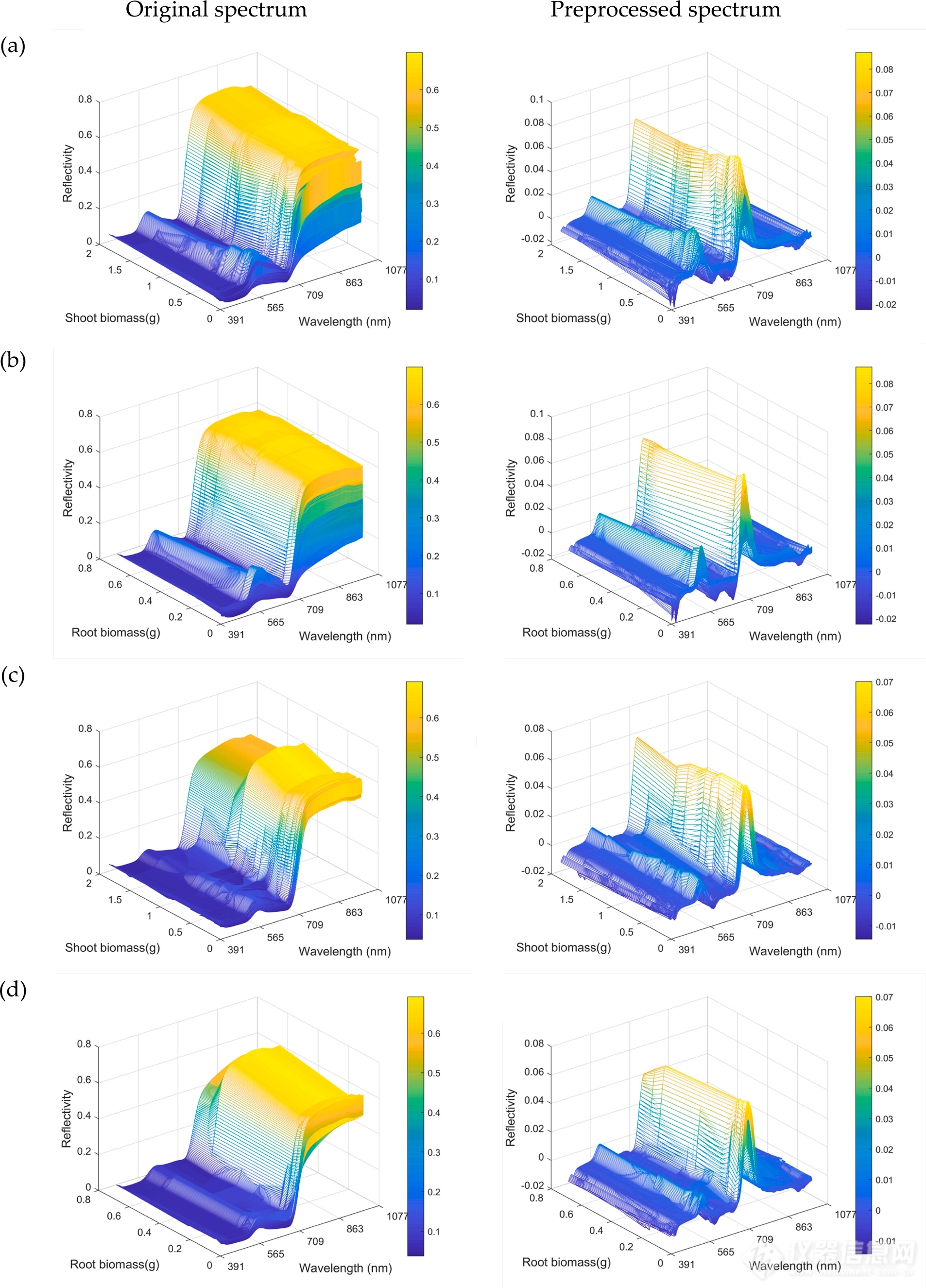
图5. 原始光谱与MSC、1D和S-G预处理后的光谱对比。(a) 茎叶光谱 + 茎叶生物量;(b) 茎叶光谱 + 根系生物量;(c) 成熟叶片光谱 + 茎叶生物量;(d) 成熟叶片光谱 + 根系生物量。
为了消除无关波段对模型准确性的影响,在基于茎叶光谱选择茎叶生物量特征波段的方法中,UVE选择的特征波段数量*多,达到212个波长,而SPA选择的特征波段数量*少,仅为8个波长;在基于茎叶光谱选择根系生物量特征波段的方法中,UVE选择的特征波段数量*多,为135个波长,SPA选择的特征波段数量*少,为6个波长;在基于成熟叶片光谱选择茎叶生物量特征波段的方法中,UVE选择的特征波段数量*多,为69个波长,SPA选择的特征波段数量*少,为14个波长;在基于成熟叶片光谱选择根系生物量特征波段的方法中,UVE选择的特征波段数量*多,为90个波长,SPA选择的特征波段数量*少,为17个波长。
在基于茎叶光谱评估茎叶生物量时,UVE算法的建模效果优于CARS和SPA,UVE+CNN-GRU提供了*佳的估算模型(Rp²=0.90,RMSEP=0.12,RPD=2.43)。CARS算法的建模效果较差,CARS+PLSR模型的效果*差(Rp²=0.50,RMSEP=0.32,RPD=1.36)。
在基于成熟叶片光谱评估茎叶生物量时,UVE算法的建模效果优于CARS和SPA,UVE+CNN-GRU提供了*佳的估算模型(Rp²=0.78,RMSEP=0.16,RPD=2.13)。SPA算法的建模效果较差,SPA+PLSR模型的效果*差(Rp²=0.48,RMSEP=0.29,RPD=1.00)。在基于成熟叶片光谱评估根系生物量时,SPA算法的建模效果优于UVE和CARS,SPA+LSTM提供了*佳的估算模型(Rp²=0.65,RMSEP=0.05,RPD=1.67)。CARS算法的建模效果较差,CARS+PLSR模型的效果*差(Rp²=0.39,RMSEP=0.10,RPD=1.22)。图6展示了四种*佳估算模型的预测值与实际值的散点图。
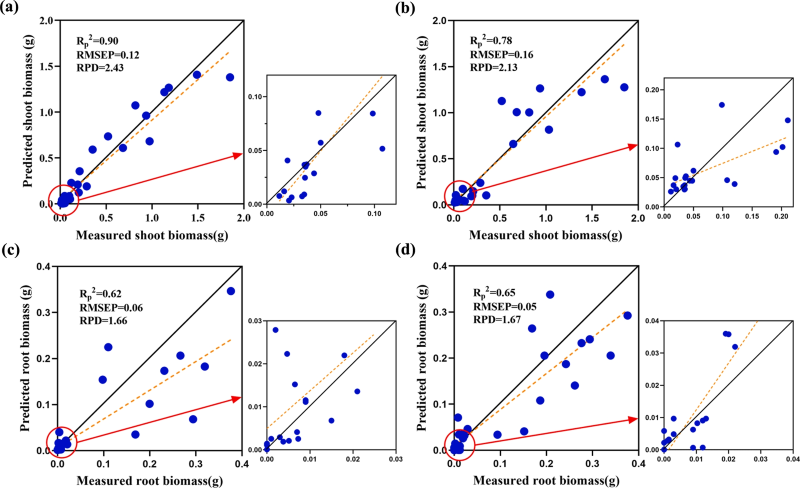
图6. 四种*佳估算模型的预测值与实际值的散点图。(a) 茎叶光谱 + UVE + CNN-GRU;(b) 成熟叶片光谱 + UVE + CNN-GRU;(c) 茎叶光谱 + SPA + CNN;(d) 成熟叶片光谱 + SPA + LSTM。
结论
本研究提出了一种利用高光谱成像技术监测茶扦插苗茎叶生长和根系生长的方法。首先,通过Mask R-CNN提取茶扦插苗成熟叶片的光谱和茎叶的光谱。随后,利用MSC、S-G滤波和1-D对光谱进行预处理,并通过UVE、CARS和SPA筛选特征波段。*后,采用CNN-GRU网络构建茎叶和根系生物量的预测模型。研究结果表明:(1)Mask R-CNN能**提取成熟叶片(**率=97.8%)和嫩梢(**率=91.5%)的光谱特征;(2)通过UVE方法筛选获得的嫩梢(212个)和根系(105个)生物量特征波段,较CARS和SPA方法更具丰富性;(3)基于嫩梢光谱构建的UVE+CNN-GRU模型(Rp²=0.90,RMSEP=0.12,RPD=2.43)对嫩梢生物量的估算效果*优,表明该模型预测结果可靠,与实际值误差较小;(4)基于成熟叶片光谱构建的SPA+LSTM模型(Rp²=0.65,RMSEP=0.05,RPD=1.67)对根系生物量的估算效果*佳,证明该模型可用于茶树扦插苗根系状况评估,为根系生长监测提供了有效手段。
